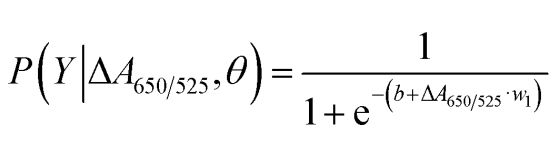Die Krebsfrüherkennung mittels Flüssigbiopsie ist eine neue Methode der Krebserkennung und -diagnose, die das US-amerikanische National Cancer Institute in den letzten Jahren vorgeschlagen hat. Ziel ist die Früherkennung von Krebs oder sogar präkanzerösen Läsionen. Die Methode wird häufig als neuartiger Biomarker für die Früherkennung verschiedener bösartiger Erkrankungen eingesetzt, darunter Lungenkrebs, Magen-Darm-Tumoren, Gliome und gynäkologische Tumoren.
Die Entstehung von Plattformen zur Identifizierung von Methylierungslandschafts-Biomarkern (Methylscape) hat das Potenzial, die bestehende Früherkennung von Krebserkrankungen deutlich zu verbessern und die Patienten in das frühestmögliche behandelbare Stadium zu bringen.
Kürzlich haben Forscher eine einfache und direkte Sensorplattform zur Erkennung von Methylierungsstrukturen entwickelt. Diese basiert auf Cysteamin-beschichteten Goldnanopartikeln (Cyst/AuNPs) in Kombination mit einem Smartphone-basierten Biosensor und ermöglicht so ein schnelles Frühscreening einer Vielzahl von Tumoren. Ein Frühscreening auf Leukämie kann innerhalb von 15 Minuten nach DNA-Extraktion aus einer Blutprobe mit einer Genauigkeit von 90,0 % durchgeführt werden. Der Artikeltitel lautet: „Schnelle Erkennung von Krebs-DNA im menschlichen Blut mithilfe von Cysteamin-beschichteten AuNPs und einem Smartphone mit maschinellem Lernen“.
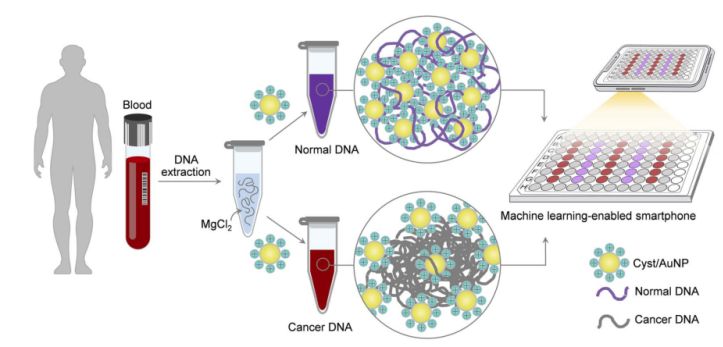
Abbildung 1. Eine einfache und schnelle Sensorplattform für das Krebs-Screening über Zysten-/AuNP-Komponenten kann in zwei einfachen Schritten erstellt werden.
Dies ist in Abbildung 1 dargestellt. Zunächst wurden die DNA-Fragmente in einer wässrigen Lösung gelöst. Anschließend wurden Cyst/AuNPs zu der gemischten Lösung hinzugefügt. Normale und maligne DNA weisen unterschiedliche Methylierungseigenschaften auf, was zu DNA-Fragmenten mit unterschiedlichen Selbstassemblierungsmustern führt. Normale DNA aggregiert lose und aggregiert schließlich Cyst/AuNPs, was zur Rotverschiebung der Cyst/AuNPs führt, sodass ein Farbwechsel von Rot nach Violett mit bloßem Auge erkennbar ist. Im Gegensatz dazu führt das einzigartige Methylierungsprofil von Krebs-DNA zur Bildung größerer Cluster von DNA-Fragmenten.
Bilder von 96-Well-Platten wurden mit einer Smartphone-Kamera aufgenommen. Die Krebs-DNA wurde mit einem Smartphone gemessen, das mit maschinellem Lernen ausgestattet war, im Vergleich zu spektroskopiebasierten Methoden.
Krebsvorsorge in echten Blutproben
Um den Nutzen der Sensorplattform zu erweitern, setzten die Forscher einen Sensor ein, der in echten Blutproben erfolgreich zwischen normaler und krebsartiger DNA unterscheiden konnte. Methylierungsmuster an CpG-Stellen regulieren die Genexpression epigenetisch. Bei fast allen Krebsarten wurden alternierende Veränderungen der DNA-Methylierung und damit der Expression von Genen beobachtet, die die Tumorentstehung fördern.
Als Modell für andere mit DNA-Methylierung assoziierte Krebsarten verwendeten die Forscher Blutproben von Leukämiepatienten und gesunden Kontrollpersonen, um die Wirksamkeit der Methylierungslandschaft bei der Differenzierung von Leukämiekrebs zu untersuchen. Dieser Methylierungslandschafts-Biomarker übertrifft nicht nur bestehende Schnellscreeningmethoden für Leukämie, sondern zeigt auch, dass sich mit diesem einfachen und unkomplizierten Test die Früherkennung einer Vielzahl von Krebsarten ausweiten lässt.
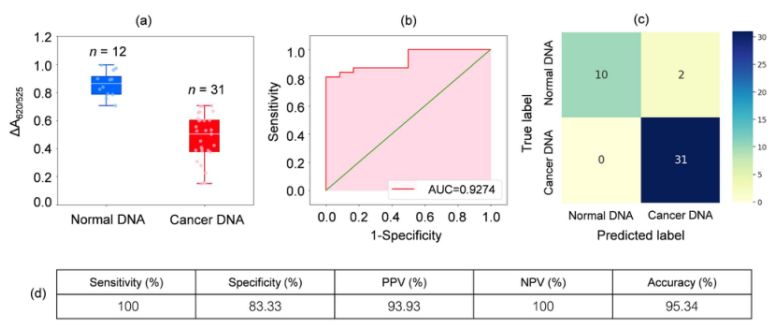
DNA aus Blutproben von 31 Leukämiepatienten und 12 gesunden Personen wurde analysiert. Wie im Boxplot in Abbildung 2a gezeigt, war die relative Absorption der Krebsproben (ΔA650/525) niedriger als die der DNA aus normalen Proben. Dies lag hauptsächlich an der erhöhten Hydrophobie, die zu einer dichten Aggregation der Krebs-DNA führte, welche die Aggregation von Cyst/AuNPs verhinderte. Infolgedessen waren diese Nanopartikel vollständig in den äußeren Schichten der Krebsaggregate dispergiert, was zu einer unterschiedlichen Dispersion der an normale und Krebs-DNA-Aggregate adsorbierten Cyst/AuNPs führte. ROC-Kurven wurden dann erstellt, indem der Schwellenwert von einem Minimalwert von ΔA650/525 bis zu einem Maximalwert variiert wurde.
Abbildung 2.(a) Relative Absorptionswerte von Zysten/AuNP-Lösungen, die das Vorhandensein von normaler (blau) und Krebs-DNA (rot) unter optimierten Bedingungen zeigen
(DA650/525) von Boxplots; (b) ROC-Analyse und Auswertung diagnostischer Tests. (c) Konfusionsmatrix zur Diagnose von Normal- und Krebspatienten. (d) Sensitivität, Spezifität, positiver Vorhersagewert (PPV), negativer Vorhersagewert (NPV) und Genauigkeit der entwickelten Methode.
Wie in Abbildung 2b dargestellt, zeigte die für den entwickelten Sensor ermittelte Fläche unter der ROC-Kurve (AUC = 0,9274) eine hohe Sensitivität und Spezifität. Wie aus dem Boxplot ersichtlich ist, ist der niedrigste Punkt, der die Gruppe mit normaler DNA darstellt, nicht deutlich vom höchsten Punkt getrennt, der die Gruppe mit Krebs-DNA darstellt. Daher wurde zur Unterscheidung zwischen der Gruppe mit normaler DNA und der Gruppe mit Krebs eine logistische Regression verwendet. Anhand einer Reihe unabhängiger Variablen wird die Wahrscheinlichkeit des Eintretens eines Ereignisses, beispielsweise einer Gruppe mit Krebs- oder normaler DNA, geschätzt. Die abhängige Variable liegt zwischen 0 und 1. Das Ergebnis ist daher eine Wahrscheinlichkeit. Wir haben die Wahrscheinlichkeit der Krebsidentifizierung (P) basierend auf ΔA650/525 wie folgt ermittelt.
wobei b=5,3533,w1=-6,965. Bei der Probenklassifizierung deutet eine Wahrscheinlichkeit von weniger als 0,5 auf eine normale Probe hin, während eine Wahrscheinlichkeit von 0,5 oder höher auf eine Krebsprobe hinweist. Abbildung 2c zeigt die Konfusionsmatrix, die aus der Leave-it-alone-Kreuzvalidierung generiert wurde und zur Validierung der Stabilität der Klassifizierungsmethode verwendet wurde. Abbildung 2d fasst die diagnostische Testbewertung der Methode zusammen, einschließlich Sensitivität, Spezifität, positivem Vorhersagewert (PPV) und negativem Vorhersagewert (NPV).
Smartphone-basierte Biosensoren
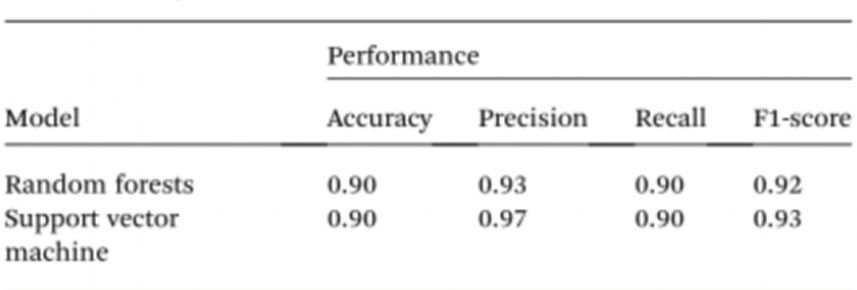
Um die Probenuntersuchung ohne den Einsatz von Spektralphotometern weiter zu vereinfachen, nutzten die Forscher künstliche Intelligenz (KI), um die Farbe der Lösung zu interpretieren und zwischen normalen und krebskranken Personen zu unterscheiden. Dazu wurde Computer Vision eingesetzt, um die Farbe der Cyst/AuNPs-Lösung anhand von Bildern von 96-Well-Platten, die mit einer Handykamera aufgenommen wurden, in normale DNA (violett) oder krebsartige DNA (rot) zu übersetzen. Künstliche Intelligenz kann die Kosten senken und die Interpretation der Farbe von Nanopartikellösungen erleichtern, und zwar ohne den Einsatz optischer Smartphone-Hardware. Schließlich wurden zwei maschinelle Lernmodelle trainiert, darunter Random Forest (RF) und Support Vector Machine (SVM), um die Modelle zu erstellen. Sowohl das RF- als auch das SVM-Modell klassifizierten die Proben mit einer Genauigkeit von 90,0 % korrekt als positiv und negativ. Dies deutet darauf hin, dass der Einsatz künstlicher Intelligenz in der Handy-basierten Biosensorik durchaus möglich ist.
Abbildung 3. (a) Zielklasse der Lösung, die während der Vorbereitung der Probe für den Bildaufnahmeschritt aufgezeichnet wurde. (b) Beispielbild, das während des Bildaufnahmeschritts aufgenommen wurde. (c) Farbintensität der Zysten-/AuNP-Lösung in jeder Vertiefung der 96-Well-Platte, extrahiert aus dem Bild (b).
Mithilfe von Cyst/AuNPs haben Forscher erfolgreich eine einfache Sensorplattform zur Erkennung der Methylierungslandschaft und einen Sensor entwickelt, der in der Lage ist, bei der Verwendung von echten Blutproben für das Leukämie-Screening normale DNA von Krebs-DNA zu unterscheiden. Der entwickelte Sensor zeigte, dass aus echten Blutproben extrahierte DNA in der Lage war, geringe Mengen Krebs-DNA (3 nM) bei Leukämiepatienten innerhalb von 15 Minuten schnell und kostengünstig zu erkennen, und zwar mit einer Genauigkeit von 95,3 %. Um die Probenuntersuchung durch den Wegfall eines Spektralphotometers weiter zu vereinfachen, wurde maschinelles Lernen eingesetzt, um die Farbe der Lösung zu interpretieren und anhand eines Handyfotos zwischen normalen und krebskranken Personen zu unterscheiden. Auch hier konnte eine Genauigkeit von 90,0 % erreicht werden.
Referenz: DOI: 10.1039/d2ra05725e
Veröffentlichungszeit: 18. Februar 2023
 中文网站
中文网站