Während der PCR-Reaktion treten häufig einige Störfaktoren auf.
Aufgrund der sehr hohen Sensitivität der PCR gilt die Kontamination als einer der wichtigsten Einflussfaktoren auf die PCR-Ergebnisse und kann zu falsch positiven Ergebnissen führen.
Ebenso kritisch sind die verschiedenen Quellen, die zu falsch-negativen Ergebnissen führen.Wenn ein oder mehrere wesentliche Teile des PCR-Gemischs oder die Amplifikationsreaktion selbst gehemmt oder beeinträchtigt werden, kann der diagnostische Test behindert werden.Dies kann zu einer verringerten Effizienz und sogar zu falsch negativen Ergebnissen führen.
Zusätzlich zur Hemmung kann es aufgrund der Transport- und/oder Lagerbedingungen vor der Probenvorbereitung zu einem Verlust der Integrität der Zielnukleinsäure kommen.Insbesondere hohe Temperaturen oder unzureichende Lagerung können zur Schädigung von Zellen und Nukleinsäuren führen.Zell- und Gewebefixierung und Paraffineinbettung sind bekannte Ursachen für DNA-Fragmentierung und ein anhaltendes Problem (siehe Abbildungen 1 und 2).In diesen Fällen hilft auch eine optimale Isolierung und Reinigung nicht.
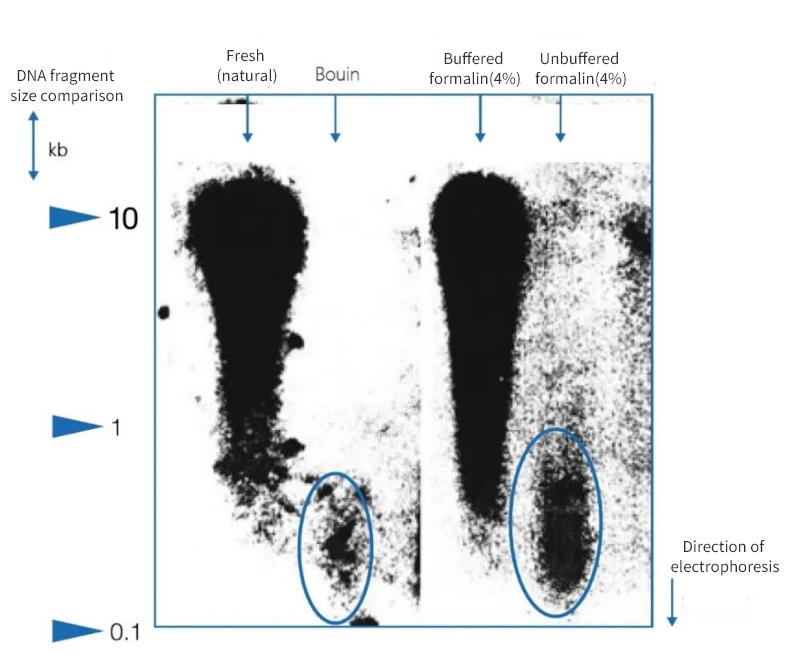
Abbildung 1 |Auswirkung der Immobilisierung auf die DNA-Integrität
Die Agarosegelelektrophorese zeigte, dass die Qualität der aus Paraffinschnitten von Autopsien isolierten DNA erheblich schwankte.Abhängig von der Fixierungsmethode war in den Extrakten DNA mit unterschiedlichen durchschnittlichen Fragmentlängen vorhanden.Die DNA blieb nur erhalten, wenn sie in nativen gefrorenen Proben und in gepuffertem neutralem Formalin fixiert wurde.Die Verwendung eines stark sauren Bouin-Fixativs oder ungepufferten, ameisensäurehaltigen Formalin führte zu einem erheblichen DNA-Verlust.Der verbleibende Anteil ist stark fragmentiert.
Auf der linken Seite wird die Länge der Fragmente in Kilobasenpaaren (kbp) ausgedrückt.
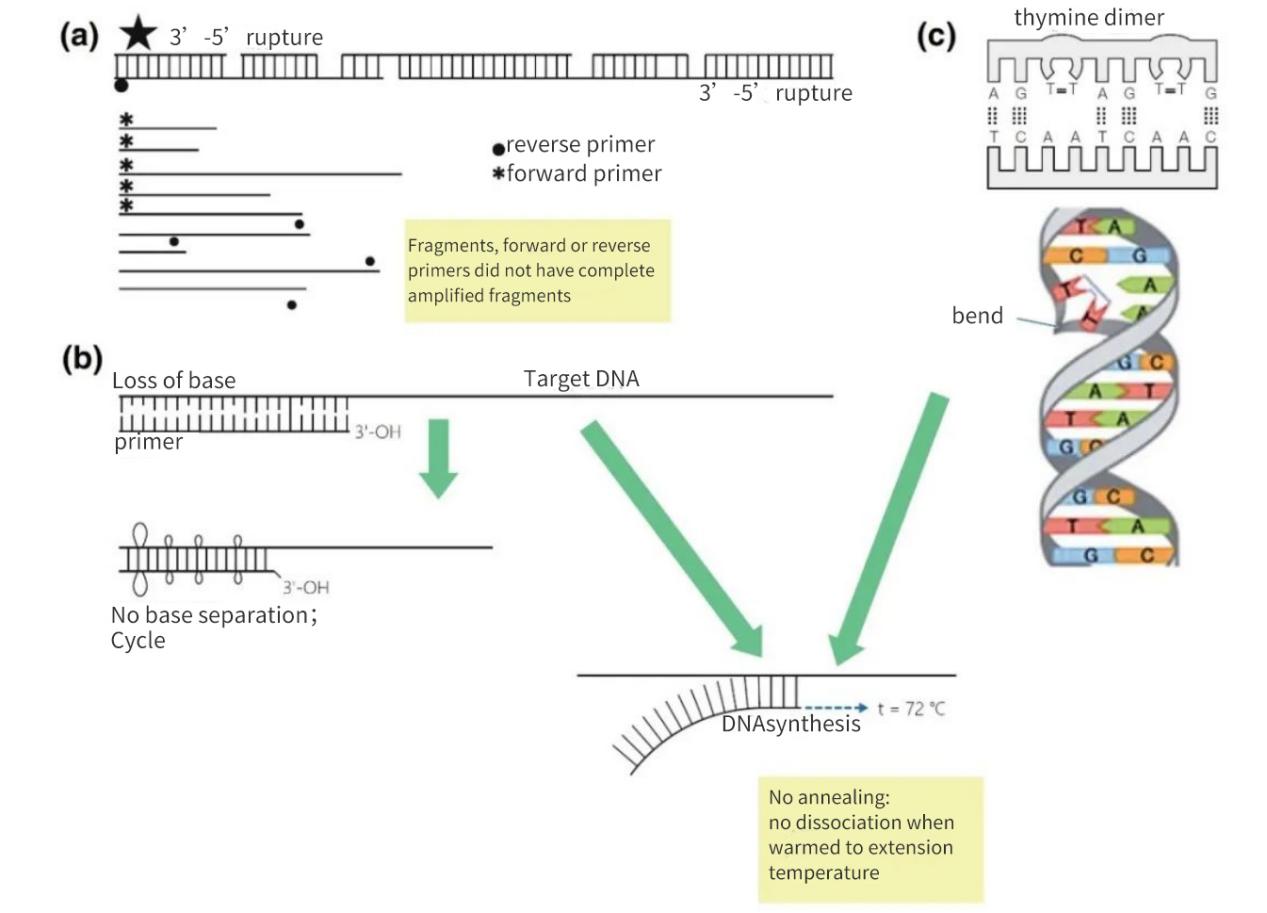
Abbildung 2 |Verlust der Integrität von Nukleinsäurezielen
(a) Eine 3′-5′-Lücke auf beiden Strängen führt zu einem Bruch in der Ziel-DNA.Die DNA-Synthese findet weiterhin auf dem kleinen Fragment statt.Fehlt jedoch eine Primer-Annealing-Stelle auf dem DNA-Fragment, findet nur eine lineare Amplifikation statt.Im günstigsten Fall können sich die Fragmente gegenseitig erneut sättigen, die Ausbeuten sind jedoch gering und liegen unter der Nachweisgrenze.
(b) Der Verlust von Basen, hauptsächlich aufgrund der Depurinierung und der Bildung von Thymidin-Dimeren, führt zu einer Verringerung der Anzahl von H-Bindungen und einer Verringerung der Tm.Während der verlängerten Erwärmungsphase schmelzen die Primer von der Matrix-DNA ab und lagern sich auch unter weniger strengen Bedingungen nicht an.
(c) Benachbarte Thyminbasen bilden ein TT-Dimer.
Ein weiteres häufiges Problem, das in der molekularen Diagnostik häufig auftritt, ist die nicht optimale Freisetzung von Zielnukleinsäuren im Vergleich zur Phenol-Chloroform-Extraktion.Im Extremfall kann dies mit falsch-negativen Ergebnissen einhergehen.Durch die Siedelyse oder den enzymatischen Verdau von Zelltrümmern kann viel Zeit gespart werden, diese Methode führt jedoch häufig zu einer geringen PCR-Empfindlichkeit aufgrund einer unzureichenden Nukleinsäurefreisetzung.
Hemmung der Polymeraseaktivität während der Amplifikation
Im Allgemeinen wird Inhibition als Sammelbegriff für alle Faktoren verwendet, die zu suboptimalen PCR-Ergebnissen führen.Im streng biochemischen Sinne ist die Hemmung auf die Aktivität des Enzyms beschränkt, dh sie reduziert oder verhindert die Substrat-Produkt-Umwandlung durch Wechselwirkung mit dem aktiven Zentrum der DNA-Polymerase oder ihrem Cofaktor (z. B. Mg2+ für Taq-DNA-Polymerase).
Komponenten in der Probe oder verschiedene Puffer und Extrakte, die Reagenzien enthalten, können das Enzym direkt hemmen oder seine Cofaktoren (z. B. EDTA) abfangen, wodurch die Polymerase inaktiviert wird und wiederum zu verringerten oder falsch negativen PCR-Ergebnissen führt.
Viele Wechselwirkungen zwischen Reaktionskomponenten und zielhaltigen Nukleinsäuren werden jedoch auch als „PCR-Inhibitoren“ bezeichnet.Sobald die Integrität der Zelle durch die Isolierung gestört wird und die Nukleinsäure freigesetzt wird, kann es zu Wechselwirkungen zwischen der Probe und der sie umgebenden Lösung und Festphase kommen.Beispielsweise können „Scavenger“ einzel- oder doppelsträngige DNA durch nichtkovalente Wechselwirkungen binden und die Isolierung und Reinigung beeinträchtigen, indem sie die Anzahl der Ziele reduzieren, die schließlich das PCR-Reaktionsgefäß erreichen.
Im Allgemeinen sind PCR-Inhibitoren in den meisten Körperflüssigkeiten und Reagenzien vorhanden, die für klinische Diagnosetests verwendet werden (Harnstoff im Urin, Hämoglobin und Heparin im Blut), Nahrungsergänzungsmitteln (organische Bestandteile, Glykogen, Fett, Ca2+-Ionen) und Bestandteilen in der Umwelt (Phenole). , Schwermetalle)
| Inhibitoren | Quelle |
| Calciumionen | Milch, Knochengewebe |
| Kollagen | Gewebe |
| Gallensalz | Kot |
| Hämoglobin | In Blut |
| Hämoglobin | Blutproben |
| Huminsäure | Boden, Pflanze |
| Blut | Blut |
| Lactoferrin | Blut |
| (Europäisches) Melanin | Haut, Haare |
| Myoglobin | Muskelgewebe |
| Polysaccharide | Pflanze, Kot |
| Protease | Milch |
| Harnstoff | Urin |
| Mucopolysaccharid | Knorpel, Schleimhäute |
| Lignin, Zellulose | Pflanzen |
PCR-Inhibitoren kommen häufiger in Bakterien und eukaryotischen Zellen, Nicht-Ziel-DNA, DNA-bindenden Makromolekülen von Gewebematrizen und Laborgeräten wie Handschuhen und Kunststoffen vor.Die Reinigung von Nukleinsäuren während oder nach der Extraktion ist die bevorzugte Methode zur Entfernung von PCR-Inhibitoren.
Heutzutage können verschiedene automatisierte Extraktionsgeräte viele manuelle Protokolle ersetzen, eine 100-prozentige Rückgewinnung und/oder Reinigung der Ziele wurde jedoch nie erreicht.Potenzielle Inhibitoren können in den gereinigten Nukleinsäuren noch vorhanden sein oder bereits ihre Wirkung entfaltet haben.Es gibt verschiedene Strategien, um die Wirkung von Inhibitoren zu reduzieren.Die Wahl der geeigneten Polymerase kann einen erheblichen Einfluss auf die Inhibitoraktivität haben.Weitere bewährte Methoden zur Reduzierung der PCR-Hemmung sind die Erhöhung der Polymerasekonzentration oder die Anwendung von Zusatzstoffen wie BSA.
Die Hemmung von PCR-Reaktionen kann durch den Einsatz einer internen Prozessqualitätskontrolle (IPC) nachgewiesen werden.
Es muss darauf geachtet werden, alle Reagenzien und anderen Lösungen im Extraktionskit, wie Ethanol, EDTA, CETAB, LiCl, GuSCN, SDS, Isopropanol und Phenol, durch einen gründlichen Waschschritt aus dem Nukleinsäureisolat zu entfernen.Abhängig von ihrer Konzentration können sie die PCR aktivieren oder hemmen.
Zeitpunkt der Veröffentlichung: 19. Mai 2023
 中文网站
中文网站 